عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
 Korea Genome Organization
وولوم 10
ایژو 4
صفحات 226
سال انتشار 2012شاپا الکترونیک: 2234-0742
Korea Genome Organization
وولوم 10
ایژو 4
صفحات 226
سال انتشار 2012شاپا الکترونیک: 2234-0742شاپا پرینت: 1598-866X 67 رفرنس دارد وبسایت مرجع
13,500 تومانشناسه فایل: 7969
- حجم فایل ورد: 138KB حجم پیدیاف: 302.1KB
- فرمت: فایل Word قابل ویرایش و پرینت (DOCx)
- تعداد صفحات فارسی: 12 انگلیسی: 8
- دانشگاه:
- Department of Nanobiomedical Science, WCU Research Center, Dankook University, Cheonan 330-714, Korea.
- ژورنال: Genomics & Informatics (1)
چکیده
از زمان تعیین توالی کل ژنوم، عناصرمتحرک (TEs) را به عنوان junk DNA می شناختند، به دلیل این که تعداد زیادی کپی و تکرار از این عناصر در ژنوم یوکاریوت ها وجود دارد. مطالعات زیادی در باره TEs برای کشف نقش آن ها در ژنوم انجام گرفته است. بر مبنای این مطالعات، می توان نقش آن ها را ایجاد تنوع ژنتیکی و ژنومی در نظر گرفت. اگرچه، نقش دقیق آن ها کاملا مشخص نشده است. عناصر متحرک از طریق مکانیسم های مختلفی تغییراتی در ژنوم میزبان خود ایجاد میکنند، مانند ورود جدید به ژنوم (de novo TE insertions)، حذف به واسطه ورود TE و همچنین نوترکیبی ژنوم. در این مقاله مروری، ما بر Alu, L1 متمرکز می شویم که یک رتروويروس اندوژن انسانی است و البته در مورد تعداد عناصر/متغیر پراکنده توالی های تکراری تاندم Alu (SVA) صحبت کرده و در مورد چگونگی تاثیر آن ها بر ژنوم پریمات ها به ویژه ژنوم انسان و شامپانزه و تنوع آن بحث خواهیم نمود.
مقدمه مقاله
عناصر متحرک (TE)، قطعاتی متحرک از ژنوم هستند که برای اولین بار، McClintok آن را کشف نمود. پس از آن، TE در تعداد زیادی از یوکاریوت ها شناسایی شد. طبق نتایج پروژه توالی یابی ژنوم، TE حدود 50% ژنوم پریمات ها را تشکیل می دهد، در حالی که DNA کدکننده تنها 2% ژنوم را تشکیل می دهد. به طور کلی TE بر اساس روش حرکت شان به دو گروه تقسیم بندی می شود: DNA ترانسپوزون و رتروترانسپوزون (تصویر 1). DNA ترانسپوزون با استفاده از مکانیسم برش و جایگذاری حرکت می –کند (cut and paste mechanism). در حالی که، رتروترانسپوزون ها به شیوه کپی و جایگذاری و دو برابر شدن عناصر در یک محل جدید از طریق RNA حدواسط جا به جا می شوند. بنابراین، تعداد کپی رتروترانسپوزون ها با سرعت بیشتری افزایش می یابد.
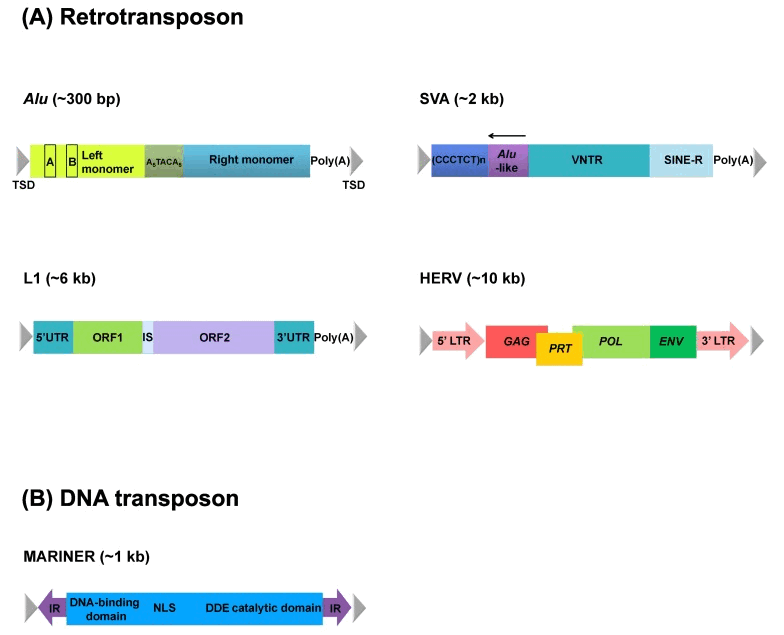
رتروترانسپوزون ها شامل عناصر پراکنده کوتاه (SINE)، عناصر پراکنده طویل (LINE) و رتروویروس آندروژن انسانی (HERV) می باشد. عناصر Alu و SVA یا تعداد متغیر عناصر پراکنده از تکرارهای تاندم Alu به گروه پریمات ها اختصاص دارد و طول کامل آن ها حدود 300 جفت باز (Alu) و 2 کیلو باز (SVA) به ترتیب می باشد. عناصر SINE بیشترین تعداد کپی را در بین عناصر Alu دارد، حدود 2/1 میلیون کپی از Alu در ژنوم انسان وجود دارد. طول LINE حدود 6 کیلو باز بوده و از SINE طویل تر است. این عناصر دو چارچوب خوانش (open reading frame) داشته که آنزیم های لازم جهت تکثیر این سه را (element) کد می کنند، انتشار عناصر Alu در ژنوم به آنزیم رونوشت بردار معکوس LINE وابسته است. SINE و LINE، تکرار های انتهایی طویل (LTR) ندارد، در حالی که HERV (حدود 10 کیلو باز) دو LTR، و سه ژن gag، pol و env بین آن ها قرار دارد.
مطالعه روی TE فعال نشان داده که احتمالا این عناصر از طریق فراهم کردن عناصر تنظیم گر cis مانند پروموتر، افزاینده (enhancer) و محل اتصال به فاکتورهای رونویسی می توانند بر بیان ژن موثر باشند (آن را تغییر دهند). از طریق این مکانیسم ها، این فعالیت تغییر یافته رونویسی می تواند منجر به ایجاد پروتئین های غیرطبیعی و غیرعملکردی شوند. از طریق مکانیسم de novo TE insertion، در ژن، عناصر TE می تواند محصولات یک ژن (پروتئین) را تغییر دهد که این تغییر می تواند برای ژنوم میزبان مفید یا مضر باشد. اگر TE وارد شده (inserted) بر ژنوم میزبان اثر منفی داشته باشد، TE به تدریج به علت موتاسیون های تکاملی و اثرات خاموش سازی غیر فعال خواهد شد.
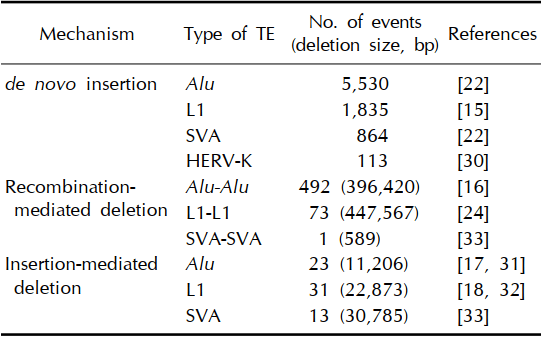
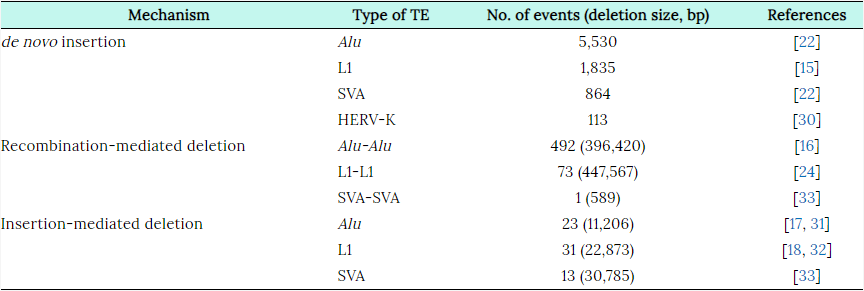
با توجه به جداسازی انسان و شامپانزه در حدود 6 میلیون سال گذشته، بسیاری از TE در ژنوم هر یک از آن ها انباشته می شود. از این عناصر موثر می توان Alu, L1, SVA و HERV-K (HML-2) را نام برد. به نظر می رسد در طول 6 میلیون سال گذشته، عناصر 5530 Alu، 1,835 L1, 864 SVA و 113 HERV-K (HML-2) جدیدا به ژنوم انسان وارد شده اند( جدول 1). این عناصر باعث ایجاد نوآرایی های اختصاصی در ژنوم انسان از طریق de novo TE insertion، حذف به واسطه ورود عناصر TE و نوترکیبی همولوگ می شود. علاوه براین، برخی از عناصری که اخیرا وارد شده اند، می توانند کپی های جدیدی در ژنوم انسان ایجاد کنند. de novo TE insertion احتمالا سبب ایجاد تفاوت های ژنومیک در جمعیت انسانی و حتی بین افراد نیز می شود که در نتیجه سببب بروز فنوتیپ و بیماری های مختلف در انسان ها می شود.
در این مقاله مروری، ما TE اختصاصی-اختصاصی را بیان کرده و در مورد اثر آن ها بر ژنوم میزبان صحبت می کنیم و مکانیسم های مورد استفاده آن ها را با ذکر مثال توضیح خواهیم داد. با توجه به همه نکات مزبور، ما معتقدیم عناصر TE که اغلب به عنوان Junk DNA معرفی شده اند، نقش های عملکردی متفاوتی داشته و نقش پویایی در تکامل ژنوم پریمات ها دارد.
ABSTRACT Transposable Elements: No More 'Junk DNA'
Since the advent of whole-genome sequencing, transposable elements (TEs), just thought to be ‘junk’ DNA, have been noticed because of their numerous copies in various eukaryotic genomes. Many studies about TEs have been conducted to discover their functions in their host genomes. Based on the results of those studies, it has been generally accepted that they have a function to cause genomic and genetic variations. However, their infinite functions are not fully elucidated. Through various mechanisms, including de novo TE insertions, TE insertion-mediated deletions, and recombination events, they manipulate their host genomes. In this review, we focus on Alu, L1, human endogenous retrovirus, and short interspersed element/variable number of tandem repeats/Alu (SVA) elements and discuss how they have affected primate genomes, especially the human and chimpanzee genomes, since their divergence.
Introduction
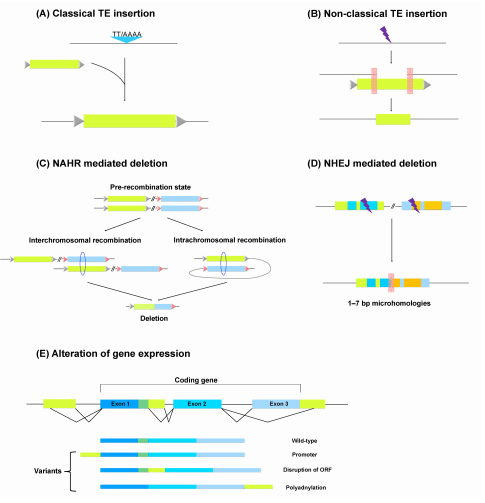
Transposable elements (TEs), mobile segments of genetic material, were first discovered by McClintock [1]. Since then, they have been identified in a variety of eukaryotes [2]. Recent genome sequencing projects have consistently shown that TEs make up ~50% of primate genomes, while coding DNA occupies only ~2% of the genomes [3-5]. TEs are generally divided into two categories, DNA transposons and retrotransposons (Fig. 1), based on their manner of mobilization. DNA transposons move using a cut-and-paste mechanism [6]. In contrast, retrotransposons move in a copy-and-paste fashion by duplicating the element into a new genomic location via an RNA intermediate [7]. Thus, retrotransposons increase their copy number more rapidly than DNA transposons.

Fig. 1 Structures of transposable elements. These elements could be categorized into retrotransposons (Alu, long interspersed element [LINE], and human endogenous retrovirus [HERV]) (A) and DNA transposons (e.g., MARINER) (B) based on their manner of mobilization. In addition, autonomous elements (e.g., HERV and LINE) have coding genes responsible for their own mobilization but also other nonautonomous elements (e.g., Alu and short interspersed element/variable number of tandem repeats/Alu [SVA]). Alu consists of two monomers separated by an A-rich connector, one of which, the left monomer, includes internal RNA polymerase III promoter (A and B boxes). Full-length of LINE is ~6 kb and has open reading frames (ORFs) encoding RNA-binding protein, endonuclease, and reverse-transcriptase, which are flanked by untraslated regions (UTRs). ORF1 and ORF2 are separated by an ~60-bp-long intergenic spacer (IS). SVA contains a (CCCTCT)n hexamer, Alu-like sequences, variable number of tandem repeat (VNTR), and short interspersed element-R (SINE-R). An arrow on Alu-like sequences indicates the direction of Alu. HERV has gag, prt, pol, and env genes flanked by a long terminal repeat (LTR), which encodes capsid protein, protease, polymerase, and envelop protein, respectively, used in viral infection. As an example of DNA transposon, mariner has a gene encoding transposase with a DNA-binding domain and catalytic domain flanked by an inverted repeat (IR). All elements are flanked by target site duplication (TSD) through integration. DDE, the conserved DDE sequence of the mariner transposase; NLS, nuclear localization signal.
Retrotranspons include short interspersed element (SINE), long interspersed element (LINE), and human endogenous retrovirus (HERV). Alu and short interspersed element/variable number of tandem repeats/Alu (SVA) elements are primate-specific retrotransposons, and their full-length is 300 bp and 2 kb, respectively. The Alu element is the most successful SINE in terms of its copy number; ~1.2 million Alu copies exist in the human genome. LINE is ~6 kb in length, and thus, it is much longer than the SINEs. This element has two open reading frames encoding enzymatic machineries essential for the propagation of the three elements; the Alu element depends on reverse transcriptase of LINE for making their dispersed copies in the host genome [8]. In contrast to SINE and LINE, which do not have long terminal repeats (LTRs), the full-length HERV (~10 kb) has two LTRs, and three genes-gag, pol, and env-are located between them [8, 9].
Studies on active TEs have suggested that the elements could alter gene expression by providing cis-regulatory elements, such as promoters, enhancers, and transcription factor binding sites [10]. Through these mechanisms, altered transcriptional activity could lead to dysfunctional and abnormal proteins. Through de novo TE insertion within a gene, TEs could alter a gene product, which could be either harmful or beneficial to its host genome [11, 12]. In cases where an inserted TE causes a harmful effect on its host genome, the TE is likely to go to inactivation and fossilization by evolutionary accumulation of mutations and silencing effects [13].
Since the divergence of the human and chimpanzee, ~6 million years ago, many TEs have propagated in each genome. Among them are the Alu, L1, SVA, and HERV-K (HML-2) elements. During the past 6 million years, 5,530 Alu, 1,835 L1, 864 SVA, and 113 HERV-K (HML-2) elements are estimated to have been newly inserted in the human genome (Table 1) [8, 14, 15]. These elements could act as an agent causing human-specific genomic rearrangements via de novo TE insertions, TE insertion-mediated deletions, and homologous recombination events [12, 16-19]. Furthermore, some of the recent integrated TEs are capable of producing new copies in the human genome. These de novo TE insertions have the potential to cause a genomic difference among human populations and even human individuals, which could be related to human phenotypes and diseases [20].
In this review, we describe species-specific TEs and discuss how they affect their host genomes, focusing on illustrating the mechanisms that they utilize with examples. Taken together, we suggest that TEs, often called ‘junk’ DNA, in fact have many functions and play a significant and dynamic role in primate genomic evolution.
- مقاله درمورد عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- بررسی عناصر متحرک (TEs) و DNA غیر مفید
- پروژه دانشجویی عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- عناصر متحرک و “DNA بی استفاده”
- پایان نامه در مورد عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- تحقیق درباره عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- مقاله دانشجویی عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان در قالب پاياننامه
- پروپوزال در مورد عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- گزارش سمینار در مورد عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان
- گزارش کارورزی درباره عناصر متحرک (TEs): نه به دیانای غیر مفید – شرح TE اختصاصی-اختصاصی و اثر آن ها بر ژنوم میزبان